Comparaison et superposition de molécules
L'activité biologique d'une molécule est souvent corrélée à sa capacité à interagir avec le site actif d'une enzyme ou bien encore au niveau d'un récepteur. Modéliser l'ensemble de l'interaction ligand-récepteur peut être relativement coûteux en temps de calcul. Parfois même si la structure 3D du récepteur n'est pas connu, rendant cette étude quasiment impossible. Dans une première approximation on peut superposer la structure 3D (préalablement optimisée) d'une molécule nouvelle avec une molécule biologiquement active et connue pour une pathologie donnée.
On peut alors savoir si la nouvelle molécule a une chance d'être active et si elle a un mécanisme d'action similaire à la molécule de référence.
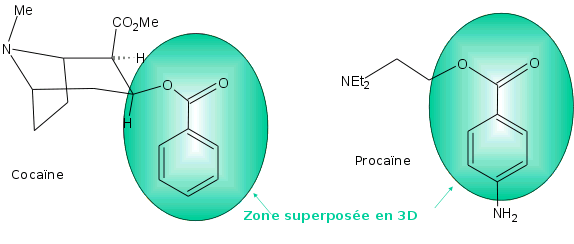
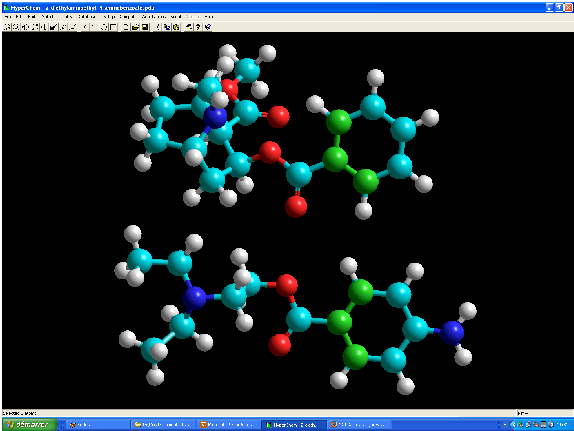
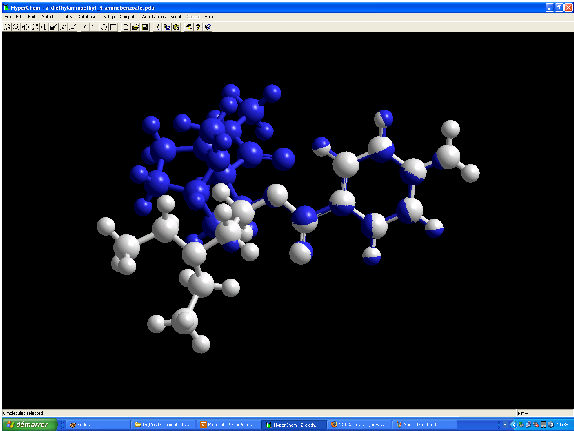
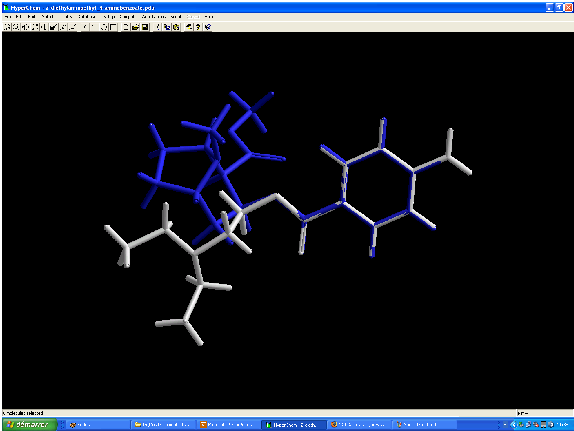
Si l'on superpose la cocaïne et la procaïne, deux anesthésiques, on constate qu'ils ont en commun la structure spatiale du cycle aromatique et de l'éther (zone spatiale importante pour expliquer l'activité sur les récepteurs concernés).
On peut également réaliser une superposition des structures pour comparer les structures électroniques ou les répartitions de charge.
Le RMS est un indice standard pour « mesurer » la « qualité » de la superposition
où
 et
et
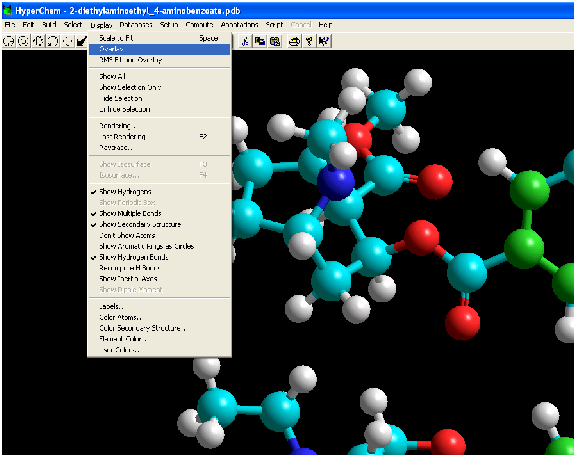
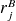 sont les coordonnées de l'atome
sont les coordonnées de l'atome
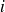 dans les molécules
dans les molécules
 et
et
 , respectivement.
, respectivement.
où
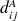 et
et
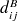 sont les distances entre les atomes
sont les distances entre les atomes
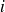 et
et
 des molécules
des molécules
 et
et
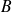 , respectivement.
, respectivement.
Au fur et à mesure que le RMS augmente on commence à voir apparaître des différences significatives entre les conformations
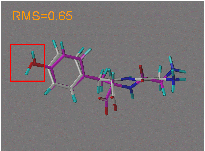
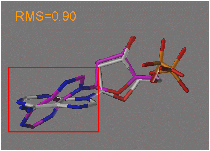

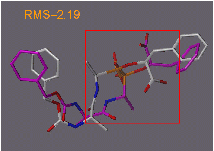
Les valeurs de RMS dépendent également de la taille des systèmes étudiés.
On peut utiliser le RMS pour superposer des protéines et étudier les différences de structure 3D entre différentes isoformes, ou encore les différences de conformation induites par une mutation.