Les interactions ligand-récepteur et la modélisation moléculaire
Plusieurs méthodes permettent d'étudier l'interaction ligand-récepteur ou encore l'interaction d'une molécule avec un site actif.
La méthode de conformation 3D est également une méthode basée sur la comparaison et la superposition.
Un pharmacophore 3D représente les positions relatives, dans l'espace, des groupes liants, au niveau d'un site actif.
Il existe en effet des distances, des angles, des orientations... caractéristiques d'une action biologique, parce que caractéristiques de l'interaction ligand/récepteur correspondante.
Sur un composé connu, on peut identifier tous les centres liants :
-
Fournisseurs ou accepteurs de liaisons hydrogène
-
Centres hydrophobes (fragments hydrocarbonés, notamment)
-
Cycles aromatiques
-
...
On (le logiciel) définit alors une « configuration spatiale idéale » à l'aide de quelques points-clef.
Un exemple bien connu est celui des antihistaminiques pour lesquels le pharmacophore 3D contient 2 cycles aromatiques + 1 azote ternaire, configuré de la manière suivante :
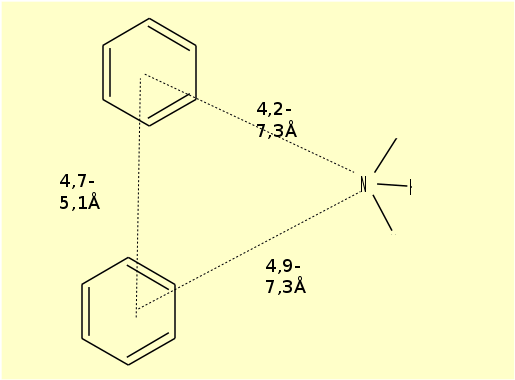
On (le logiciel) peut alors vérifier si une nouvelle molécule respecte ce pharmacophore 3D pour prédire son activité.
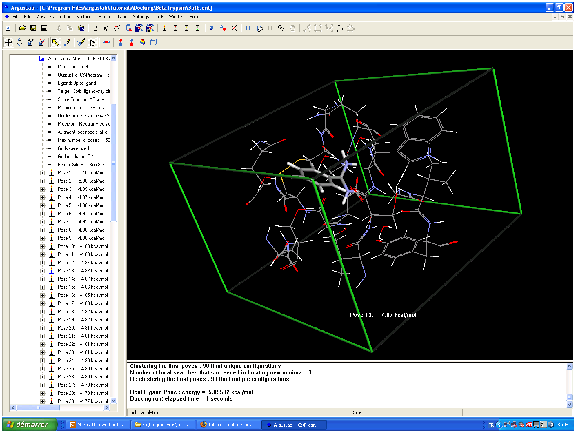
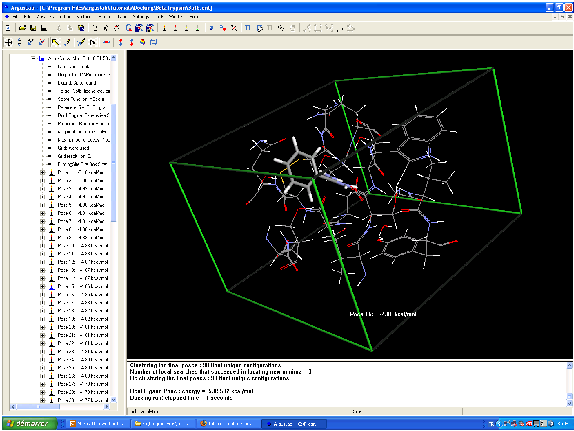
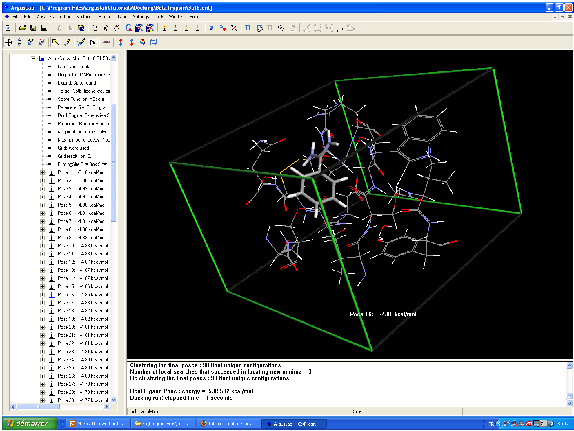
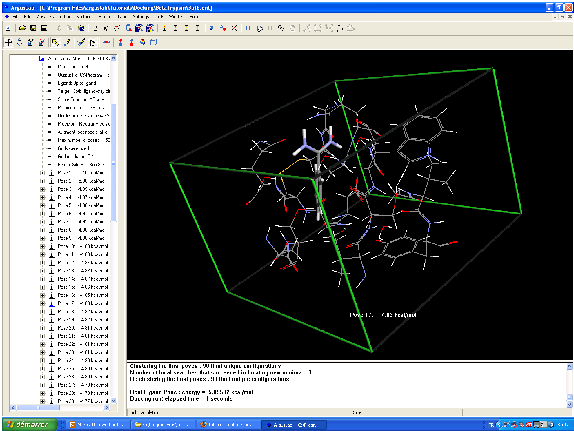
Le « docking » moléculaire consiste à prédire la ou les structures des complexes formés entre une molécule active et une protéine.
Les algorithmes de calcul génèrent aléatoirement un grand nombre d'orientations possibles pour trouver « la meilleure façon d'insérer » la molécule dans une protéine (au niveau du récepteur ou du site actif).
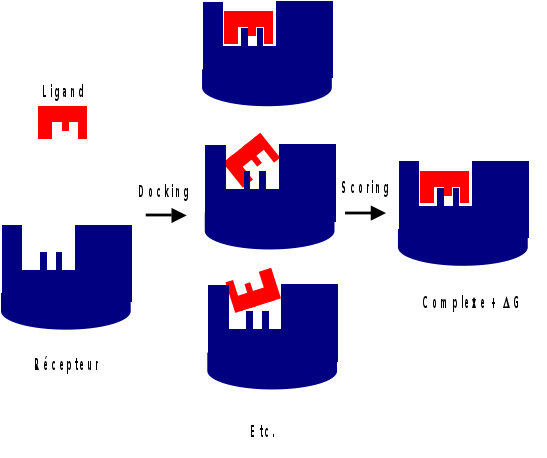
Le programme prend en compte tous les degrés de liberté de la molécule (translation et rotation).
Pour chaque possibilité l'énergie est calculée en Mécanique Moléculaire, prenant ainsi en compte toutes les interactions ligand-récepteur (liaisons de Van der Waals, liaisons H, hydrophobie...).
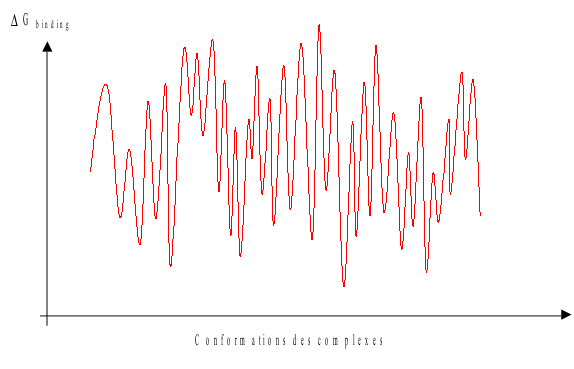
On obtient ainsi un « score » pour estimer la meilleure interaction ligand/récepteur (binding).
Le score est l'enthalpie libre de liaison (il doit être minimisé) :
En pratique, on peut obtenir (e.g., site Web de la PDB (http://www.rcsb.org/pdb/) ) la protéine complexée avec un ligand de référence.
On effectue un calcul en MM (avec un champ de force adapté).
On remplace alors le ligand de référence par la nouvelle molécule et on effectue une nouvelle optimisation de géométrie.
En comparant les scores on peut prévoir ou confirmer qu'une molécule est active.
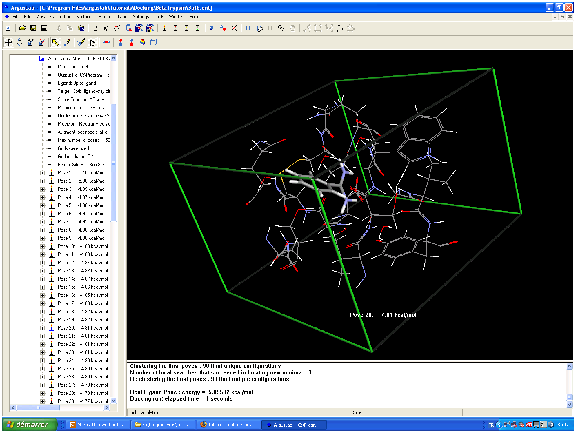
Interactions de la quercétine dans la quercétinase 2,3-dioxygénase (Thèse Sébastien Fiorucci – Nice 2006)
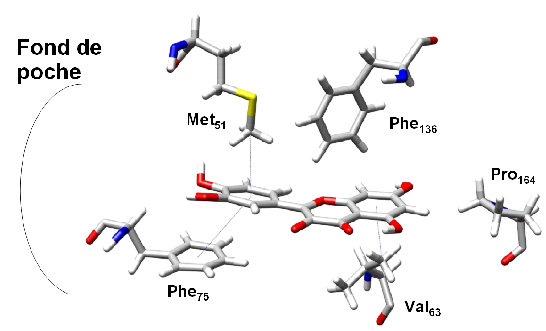
On peut également voir les modifications de conformations engendrées sur la molécule.
Pour aller plus loin (obtenir une meilleure description de l'interaction ligand/récepteur), on peut utiliser des méthodes QM/MM:
-
Utiliser des méthodes QM (ab initio ou DFT) pour le site actif et la molécule
-
Utiliser des méthodes MM pour le reste de la protéine.
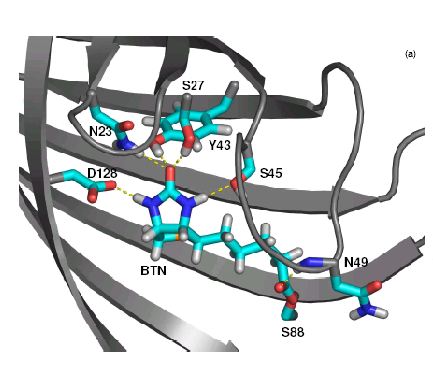
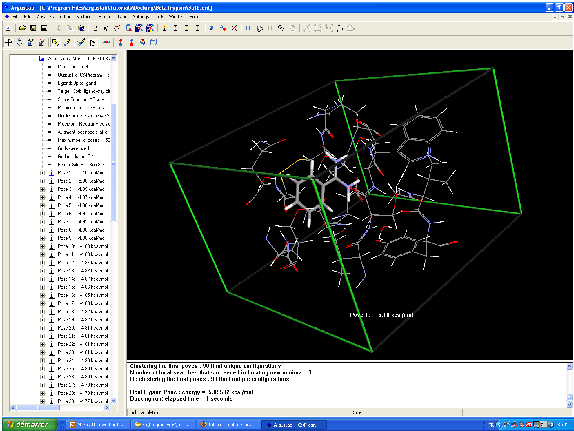
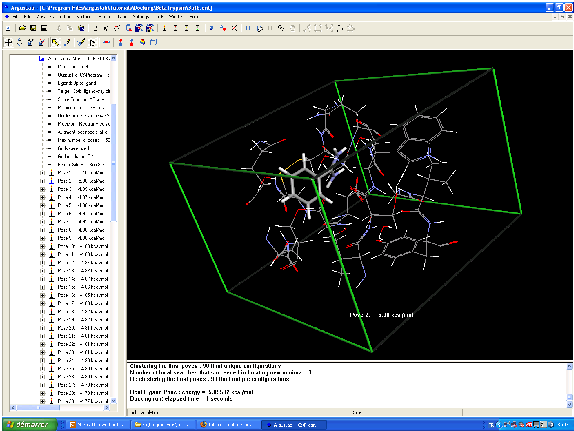
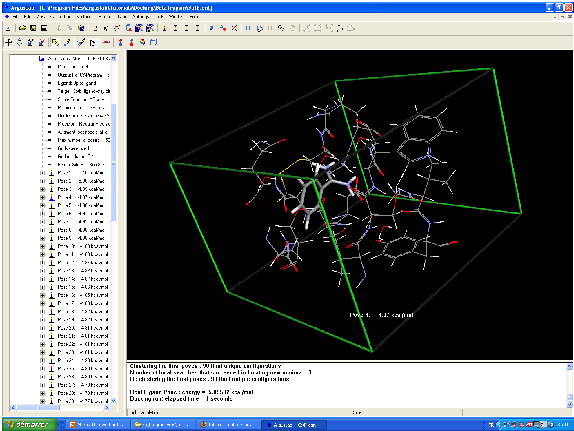
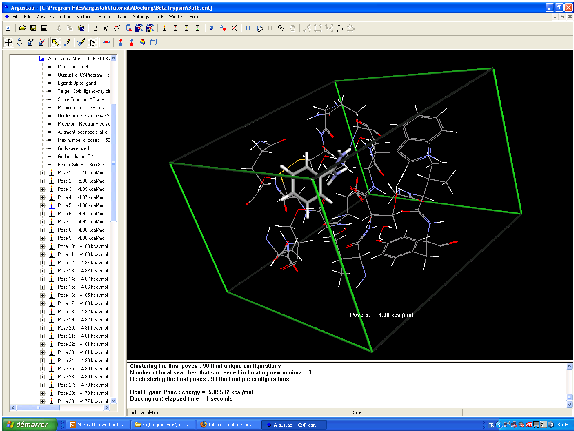
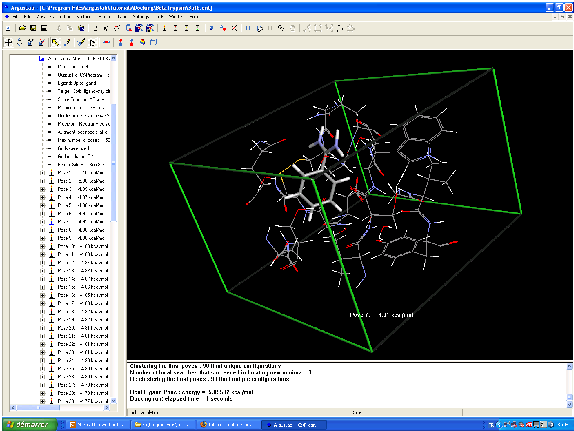
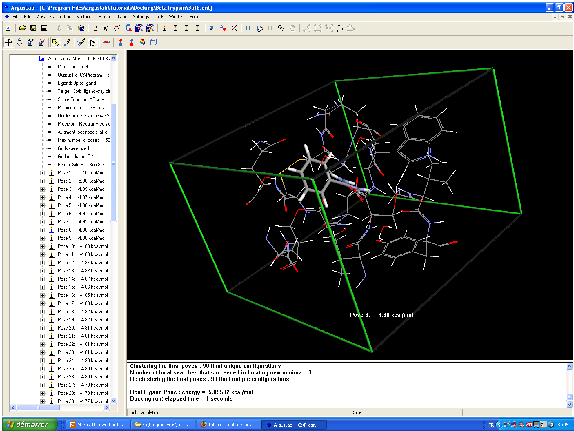
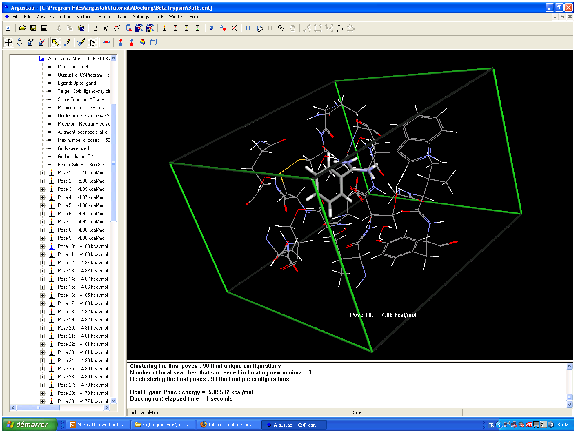
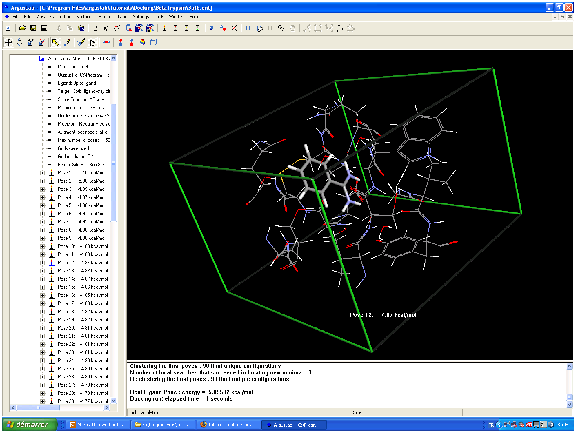
Modélisation moléculaire de la reconnaissance de la biotin par la streptavidin.
Les résidus Tyr43, Asn23, Ser27, Ser45, Asn49, Asp128 et la molécule sont calculés par une méthode ab initio.
Le calcul a notamment mis en évidence l'importance des ponts hydrogène, notamment avec les résidus Ser27, Tyr43 and Asn23