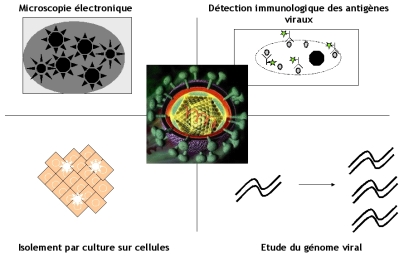
Diagnostic direct
Le diagnostic indirect a pour objectif de mettre en évidence un virus entier ou un de ses constituants (protéines virales, génome viral). Dans ce cours, nous développerons des méthodes d'isolement viral en culture cellulaire, de détection immunologique de protéines virales et de détection de génomes viraux.
Isolement viral par culture cellulaire
Ce sont les méthodes de référence pour mettre en évidence des virus. Elles reposent sur le fait qu'un virus mis en contact avec une cellule en culture peut s'y multiplier. Il faut donc disposer :
-
de cellules en culture
-
de virus se multipliant sur des cellules en culture
Cellules en culture utilisés en virologie
Le préalable à cette partie est qu'il n’existe pas de système cellulaire universel : un système cellulaire sur lequel tous les virus seraient capables de se multiplier.
En virologie, on utilise trois types de cellules en culture :
-
des cellules primaires issues directement d'organes humains ou animaux
-
des lignées continues : ce sont des cellules humaines ou animales immortalisées souvent d'origine cancéreuses qui se multiplient presque indéfiniment in vitro
-
des fibroblastes embryonnaires humains.
Afin de conserver des cellules en culture, il faut :
|
Les cellules en culture sont observées au microscope optique inversé. Elles forment un tapis cellulaire homogène confluent et adhérent. Certaines cellules poussent en suspension, on parle de cellules non adhérentes. |
Multiplication de virus en culture cellulaire
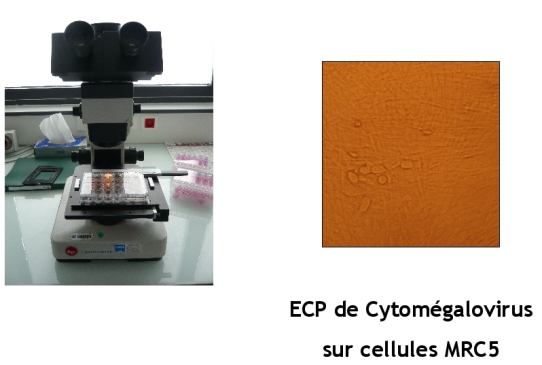
Lorsqu'on cherche à isoler un virus par culture cellulaire, le prélèvement doit être réalisé dans ce but : il doit contenir des cellules infectées et être acheminé rapidement au laboratoire. Après inoculation sur des cellules en culture, on cherche à détecter un effet cytopathogène (ECP) caractérisé par l'ensemble des altérations cytologiques observées lors de la réplication virale.
L'ECP dépend de la lignée cellulaire ensemencée et du virus qui se réplique. Il s'observe à l'état frais au microscope, à faible grossissement.
Intérêts et limites de l'isolement viral par culture cellulaire
-
L’apparition d’un ECP oriente vers une famille de virus mais doit souvent être complétée par un test spécifique pour typer le virus.
-
Ce sont les seules techniques permettant d'obtenir des particules virales afin de faire d'autres tests plus approfondis.
-
Ce sont des techniques longues qui nécessitent un grand savoir-faire de la part de techniciens de laboratoires spécialisés.
Ce sont en général des techniques peu coûteuses en terme de réactifs mais qui nécessitent un équipement de laboratoire particulier
-
Ces méthodes sont très dépendantes de la qualité du prélèvement initial qui doit absolument contenir des cellules infectées.
-
Certains virus ne donnent pas d’ECP au cours de leur réplication en culture cellulaire : virus grippaux …
-
Certains virus ne se répliquent pas en culture cellulaire : virus des hépatites, rubéole etc etc.
Détection immunologique des antigènes viraux
-
Deux techniques à connaître :
-
immunofluorescence (ou immunoperoxydase)
-
ELISA recherchant des antigènes viraux solubles
-
Immunofluorescence indirecte
Principe
L'immunofluorescence indirecte est basée sur la détection de protéines virales (antigènes) directement dans un échantillon grâce à des anticorps monoclonaux couplés à la flourescéine.
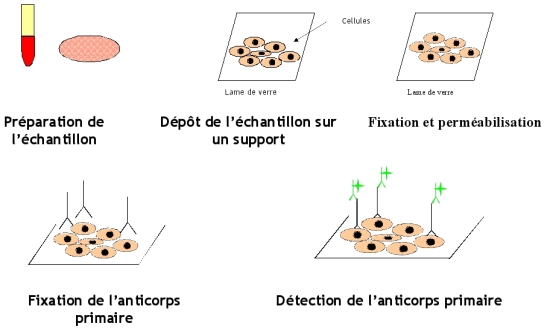
Exemples d'applications utilisées en virologie.
-
Recherche des éléments viraux directement dans l’échantillon : la recherche d'herpes simplex virus de type 2, responsable entre autres d'infections génitales vésiculeuses, peut se faire par IF à partir d'un écouvillonnage de lésions vésiculaires.
-
Identification de virus en culture cellulaire : certains virus de forment pas d'ECP comme par exemple les virus grippaux. On peut donc rechercher des antigènes viraux dans des cellules en culture 48 à 72h après l'inoculation des cellules.
-
Typage de virus d'une même famille présentant un ECP identique : les Herpes simplex virus de type 1 ou 2
Intérêts et Inconvénients des méthodes d'immunofluorescence
-
Ce sont des techniques assez spécifiques mais relativement peu sensibles.
-
Elles sont très rapides à réalisées puisqu'on peut obtenir un résultat en 3-4 heures en fonction des techniques.
-
Ces techniques sont encore une fois très dépendnates de la qualité du prélèvement : présence de cellules infectées, acheminement rapide, nécessité parfois d'utiliser un milieu de transport
-
Aucun automatisation n'est possible pour le moment.
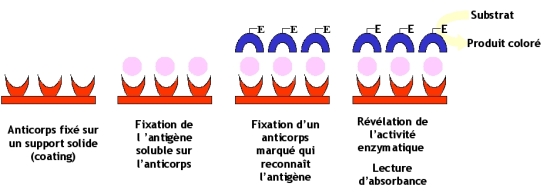
Recherche d'antigènes viraux solubles par ELISA
Au cours des infections virales, certaines protéines virales sont libérées dans le sang. On peut avoir besoin de détecter ces «antigènes solubles» présents dans le sang pour la prise en charge des patients. Dans cette application de l’ELISA, un anticorps antiviral (Ac. de capture) est fixé sur un support solide (voir schéma). Le principe général du test ELISA reste le même. Il existe de nombreux tests automatisés ou des test unitaires utilisant cette application d'ELISA.
-
Recherche des antigènes HBs et Hbe du virus de l’hépatite B :
-
Présence de l'antigène HBs dans le sang = infection en cours
-
Présence de l'antigène HBe dans le sang = réplication virale
-
Etude des génomes viraux
Nous allons maintenant exposer différentes méthodes qui permettent de détecter ou de caractériser ds génomes viraux.
-
Détection et/ou quantification de génomes viraux
-
Typage des génomes viraux
-
Recherche de mutations génomiques induisant des résistances aux traitements
Détection et/ou quantification de génomes viraux
Les outils de la biologie moléculaire ont permis l'amélioration du diagnostic des infections virales et la découverte de nouveaux virus. En effet, grâce à des outils tels que la( RT-)PCR ou le séquençage, on peut maintenant faire face à des situations de diagnostiques nouvelles. On peut :
-
Mettre en évidence la présence de génomes de virus indétectables par d’autres méthodes :
-
Recherche des papillomavirus humains (HPV) responsables de cancers du col utérin
-
Détection des virus des hépatites
-
-
Fournir une réponse rapide :
-
Recherche du génome du virus de l'herpès (HSV) au cours d'une encéphalite virale
-
-
Quantifier un génome viral :
-
Suivi des infections par le VIH ou le VHC
-
PCR en temps réel
Depuis une dizaine d'années, la PCR en temps réel a progressivement remplacé la PCR traditionnel dans les laboratoires de diagnostic en virologie. Cette technologie présente de nombreux avantages :
-
Elle est basée sur la PCR, l'existence de fluorophores et d'automates capables de lire une fluorescence
-
Elle est plus rapide et plus sensible que la PCR traditionnelle
-
Il existe différents automates ayant des spécificités différentes.
Sur les images suivantes, on voit un appareil de PCR en temps réel en microplaque. L'autre photo montre une chaîne complète permettant d'extraire les acides nucléiques, de préparer les mélanges réactionnels, de répartir les acides nucléiques et les témoins de réaction et enfin de réaliser l'amplification. |
La détection d'acides nucléiques par PCR suit différentes étapes :
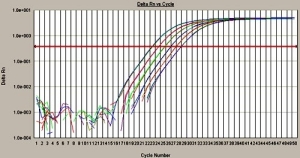
L'image montre les résultats obtenus lors d'une PCR en temps réel en technique 5'nucléase. On voit l'apparition de la fluorescence en fonction du nombre de cycles de PCR. Ces résultats sont disponibles dès la fin de la PCR sans nécessité de révélation des produits de PCR par électrophorèse en gel d'agarose ou hybridation de sondes par exemple. |
Typage des génomes viraux
Au cours de certaines infections virales, on sait que le type viral influence l'efficacité des traitements. C'est principalement le cas dans les infections par le virus de l'hépatite C. C'est une infection chronique qu'on peut traiter efficacement par l'association interféron-pégylé + ribavirine. Seulement, l'efficacité des traitements est fonction du génotype du virus :
-
Les VHC de type 1 et 3 sont moins sensibles au traitement,
-
Les VHC de type 2 et 4 sont très sensibles au traitement.
Avant de débuter un traitement contre le VHC, on réalise un typage du virus. Certaines régions du génomes viral présentent des séquences nucléotidiques variables dont les variations sont spécifiques d'un type viral. Les résultats attendus sont : VHC de type 1, 2, 3... et de sous-type a, b ou c...
Le typage du virus de l'hépatite C s'appuie de plus en plus sur le séquençage mais d'autres techniques sont disponibles telles l'hybridation sur membrane.

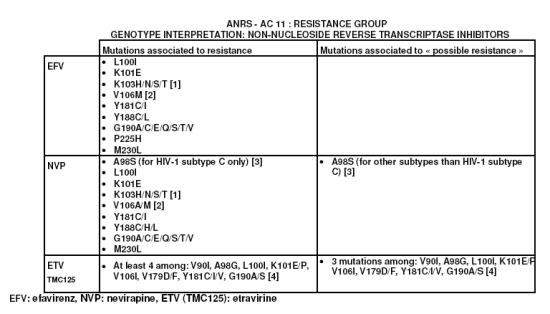
Mutations de résistance aux antiviraux
Au cours des traitements antiviraux peuvent apparaître des mutations génomiques qui induisent des résistances aux traitements. On a désormais accès à des outils de séquençage qui permettent de recherche ces mutations et donc d'adapter le traitement.
La recherche de mutations de résistance est utile pour suivre l'efficacité de tratements contre le virus de l'hépatite B (VHB), du virus de l'immunodéficience humaine (VIH) ou du cytomégalovirus (CMV)
Les traitements utilisés dans l’infection par le VIH ciblent certains protéines virales principalement :
-
La protéase virale avec les inhibiteurs de protéase
-
La transcriptase inverse avec les inhibiteurs de la transcriptase inverse
Au cours d'un traitement antirétroviral utilisant ces molécules, des mutations apparaissent sur le génome viral permettant un échappement du virus aux traitements. Nous pouvons donc séquencer les gènes codant la protéase virale ou la transcriptase inverse afin de rechercher ces mutations.
Les résultats sont rendus sous la forme suivante :
-
Résistance
-
Résistance possible
-
Absence de résistance évidente.
L'image suivante vous montre un exemple de résultats tels qu'il sont rendus par le laboratoire de virologie du CHU de Lille.
Intérêts et inconvénients des outils de biologie moléculaire en virologie.
Les différents outils que nous venons de voir présentent de nombreux avantages :
-
la grande sensibilité des techniques de PCR en temps réel permet de détecter des génomes viraux même en faible quantité dans un échantillon,
-
la quantification des génomes viraux permet de suivre finement l'efficacité d'un traitement,
-
le séquençage permet de mettre en évidence des mutations de résistances et donc d'adapter des traitements,
-
la rapidité de rendu des résultats permet de faire face à de nombreuses situations d'urgence.
Cependant, il existe de nombreux inconvénients :
la mise en évidence d'un génome viral dans un échantillon ne signifie pas que les particules virales infectieuses complètes soient présentes...
la grande sensibilité de ces techniques les rend très sensibles aux contaminations par des acides nucléiques extérieurs à l'échantillon initial,
ces technologies (PCR, PCR en temps réel, séquençage...) sont coûteuses et réservées à des laboratoires spécialisés disposant des équipements et de personnels spécialement formés.